Cap. 12 - Diversidade Taxônomica
# Pacotes necessários
library(devtools)
library(ecodados)
library (vegan)
library(ggplot2)
library(BiodiversityR)
library(hillR)
library(betapart)
library(GGally)12.1
Carregue os dados - anuros_composicao - que está no pacote ecodados. Este conjunto de dados representa a abundância de 211 espécies de anuros coletados em 44 localidades na Mata Atlântica. Calcule a riqueza de espécies para cada comunidade e os índices de Margalef, Menhinich, Shannon-Wiener, Gini-Simpson e Equitabilidade de Pielou. Salve todos os resultados em novo data frame. Faça uma gráfico usando o função ggpairs para ver a correlação entre as métricas. Qual a sua interpretação sobre os resultados?
Solução:
# Carregar a planilha com os dados
exercicio_1 <- ecodados::anuros_composicao
# Transpor o data frame para as espécies ficarem nas colunas e as localidades nas linhas
exercicio_1_t <- data.frame(t(exercicio_1))
## Calculando a riqueza observada de espécies para cada comunidade
Riqueza <- specnumber(exercicio_1_t)
## Calculando o Índice de Margalef para cada comunidade
Margalef <- round((Riqueza - 1)/log(apply(exercicio_1_t, 1, sum)), 2)
## Calculando o Índice de Menhinick para cada comunidade
Menhinick <- round(Riqueza/sqrt(apply(exercicio_1_t, 1, sum)), 2)
## Calculando o Índice de Shannon-Wiener para cada comunidade
Shannon <- round(diversity(exercicio_1_t, index = "shannon", MARGIN = 1), 2)
## Calculando o Índice de Gini-Simpson para cada comunidade
Gini_Simpson <- round(diversity(exercicio_1_t, index = "simpson", MARGIN = 1), 2)
## Calculando o Índice de Equitabilidade de Pielou para cada comunidade
Pielou <- round(Shannon/log(Riqueza), 2)
# Criando data frame
resultados <- data.frame(Riqueza, Margalef, Menhinick, Shannon, Gini_Simpson, Pielou)
# Gráfico
ggpairs(resultados, upper = list(continuous = wrap("cor", size = 4))) +
tema_livro()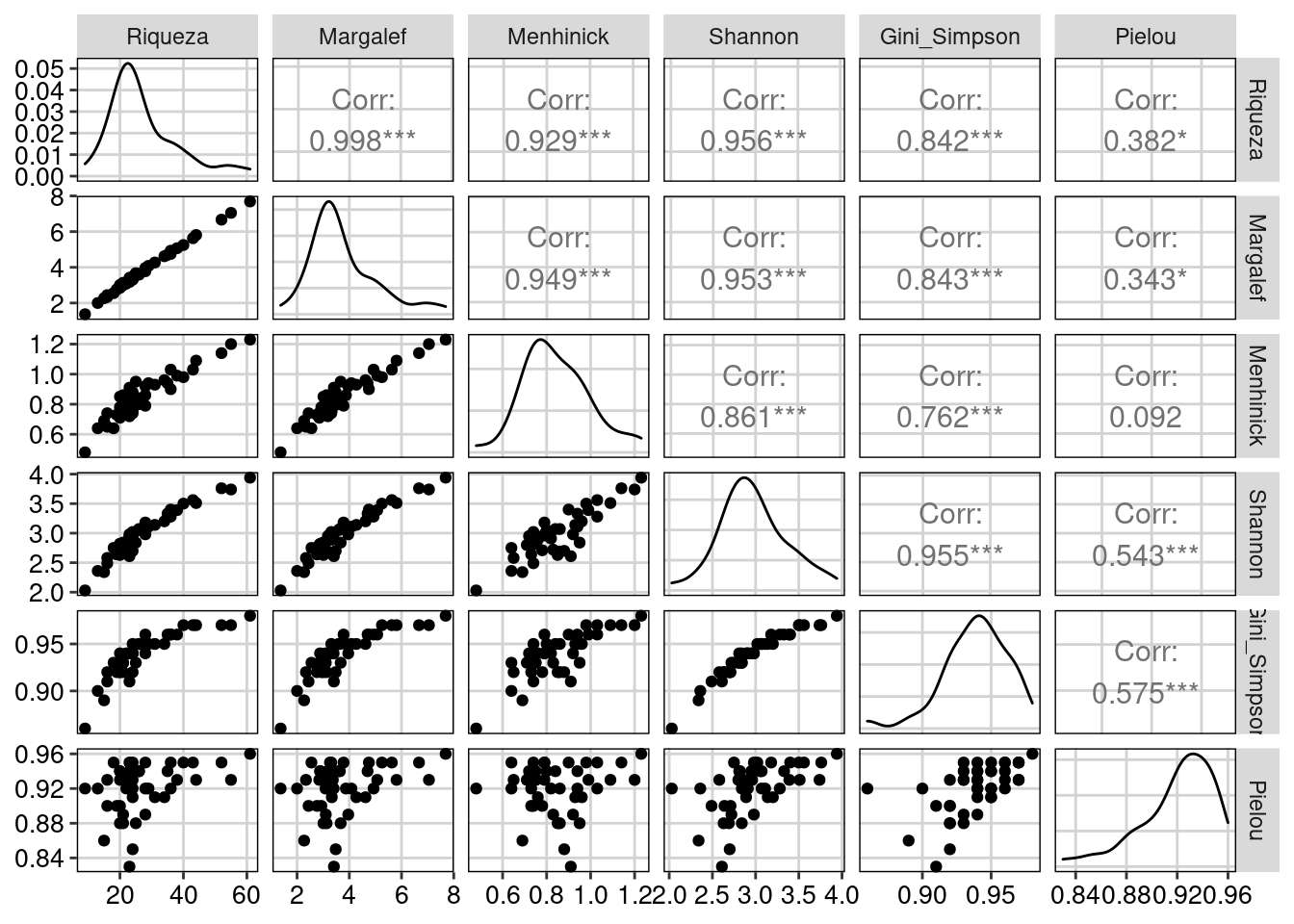
12.2 Usando os resultados anteriores, selecione as duas comunidades com os maiores e menores valores de Shannon-Wiener. Em seguida, faça um Diagrama de Whittaker. Por fim, interprete as curvas considerando as curvas teóricas (i.e., geométrica, broken-stick, etc.) descritas nos livros de ecologia.
Solução:
# Usando os resultados no exercício anterior, vamos verificar quais são as comunidades com maiores e menores valores de Shannon-Wienner
order(resultados$Shannon)
#> [1] 43 37 12 4 41 1 16 38 19 11 22 44 20 13 10 29 17 24
#> [19] 42 8 15 25 31 7 5 14 18 3 21 39 40 32 30 34 33 27
#> [37] 36 9 2 35 26 6 28 23
# AS comunidades com maiores valores estão nas linhas 6 e 23 e as comunidades com menores valores nas linhas 43 e 37.
rank_com6 <- rankabundance(exercicio_1_t[6, exercicio_1_t[6,] > 0])
rank_com23 <- rankabundance(exercicio_1_t[23, exercicio_1_t[23,] > 0])
rank_com43 <- rankabundance(exercicio_1_t[43, exercicio_1_t[43,] > 0])
rank_com37 <- rankabundance(exercicio_1_t[37, exercicio_1_t[37,] > 0])
## Gráfico
rankabunplot(rank_com6, scale = "logabun", pch = 19, specnames = NULL,
col = "darkorange")
rankabunplot(rank_com23, scale = "logabun", pch = 19, specnames = NULL,
addit = TRUE, col = "red")
rankabunplot(rank_com43, scale = "logabun", pch = 19, specnames = NULL,
addit = TRUE, col = "cyan4" )
rankabunplot(rank_com37, scale = "logabun", pch = 19, specnames = NULL,
addit = TRUE, col = "darkblue" )
legend(20, 8, legend = c("Comunidade 6", "Comunidade 23",
"Comunidade 43", "Comunidade 37"),
col = c("darkorange", "red", "cyan4", "darkblue"), lty = 1, lwd = 3,
cex = 1.2, box.lty = 0)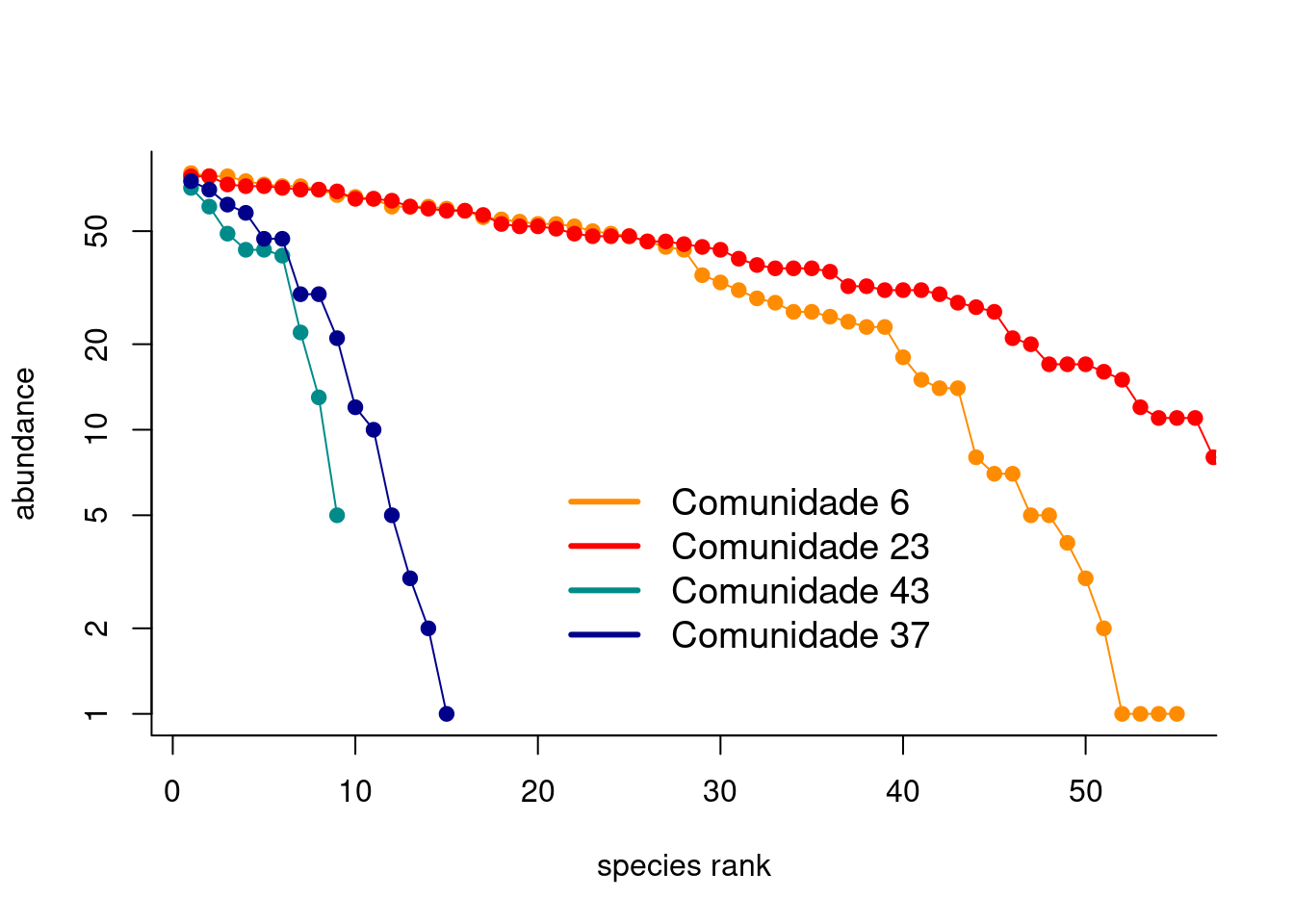
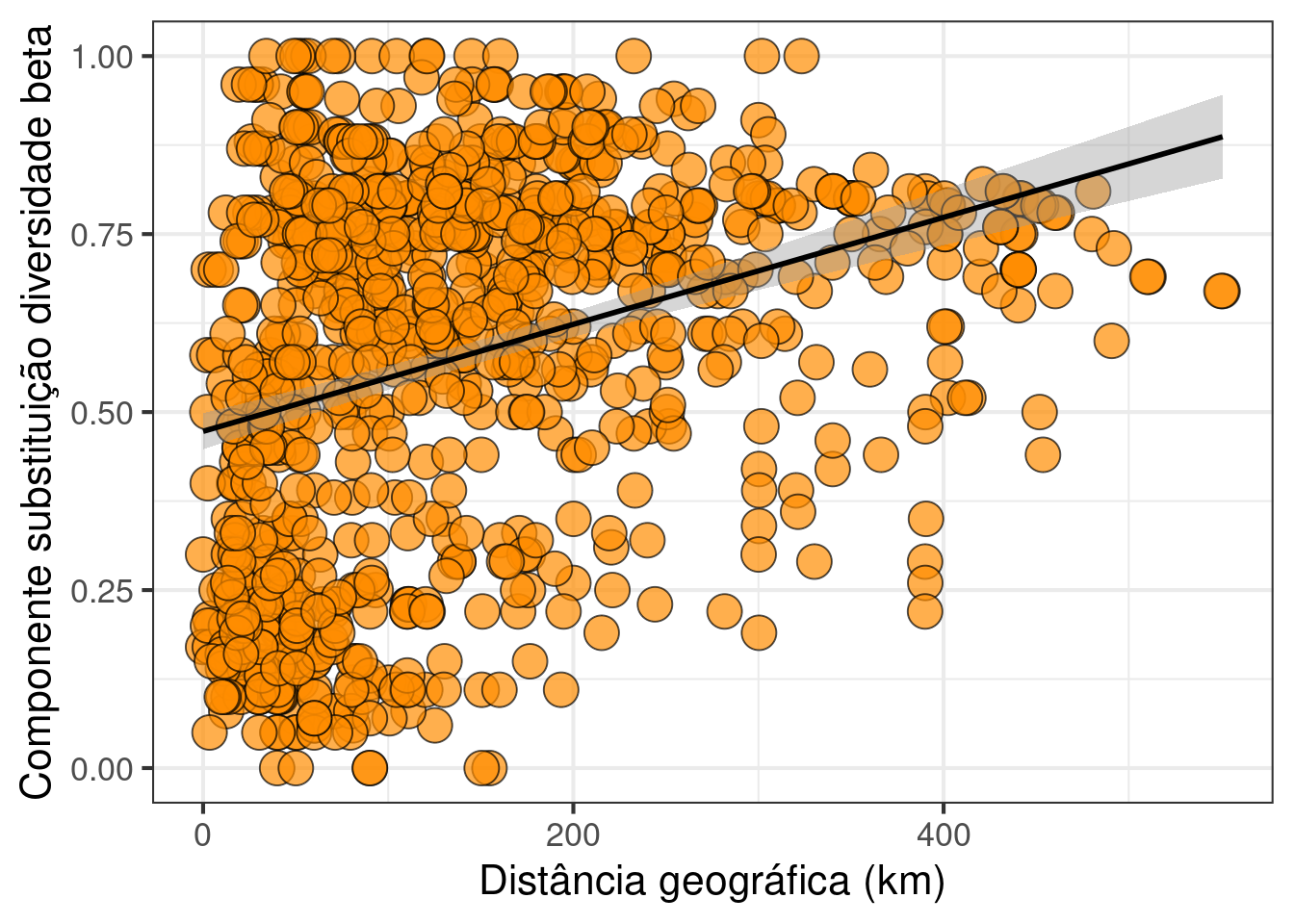
12.3 Usando os dados - anuros_composicao - calcule a partição da diversidade beta considerando os dados de abundância e presença e ausência. a) Faça um gráfico boxplot com os resultados. Discuta se os resultados usando abundância ou presença e ausência são congruentes ou discrepantes. b) Calcule a distância geográfica (use a planilha anuros_ambientais) entre as localidaes (use a Distância euclidiana). Em seguida, faça uma análise de regressão para verificar se as localidades que estão próximas apresentam maior similaridade na composição de espécies (use componente turnover - Bsim) do que as comunidades que estão distantes (e.g., Decaimento da similaridade).
Solução:
# Carregar a planilha com os dados
anuros <- ecodados::anuros_composicao
# Transpor o data frame
anuros_t <- data.frame(t(anuros))
## Transformando dados em presencia e ausência.
anuros_PA <- decostand(anuros_t, method = "pa")
## Diversidade beta presença e ausência
resultado_PA <- beta.pair(anuros_PA, index.family = "sorensen")
## Diversidade beta abundância
resultado_Abund <- beta.pair.abund(anuros_t, index.family = "bray")
## Criando data frame com os resultados
diver_beta <- c(round(as.numeric(resultado_PA$beta.sor), 2),
round(as.numeric(resultado_PA$beta.sim), 2),
round(as.numeric(resultado_PA$beta.sne), 2),
round(as.numeric(resultado_Abund$beta.bray), 2),
round(as.numeric(resultado_Abund$beta.bray.bal), 2),
round(as.numeric(resultado_Abund$beta.bray.gra), 2))
Componentes <- rep(c("Bsor", "Bsim", "Bnes", "BBray", "BBray_bal", "BBray_gra"),
each = 946)
Dados <- rep(c("Incidencia", "Abundância"),
each = 2838)
data_frame <- data.frame(diver_beta, Componentes, Dados)
# Reordenando os componentes para melhorar visualização no gráfico
data_frame$Componentes <- factor(data_frame$Componentes , levels=c("Bsor", "Bsim", "Bnes",
"BBray", "BBray_bal", "BBray_gra"))
## Gráfico
ggplot(data_frame, aes(y = diver_beta , x = Componentes, fill = Dados)) +
geom_boxplot() +
theme_bw(base_size = 16) +
geom_jitter(size = 0.3, width = 0.05) +
labs( x = "Componentes diversidade beta", y = "Dissimilaridade")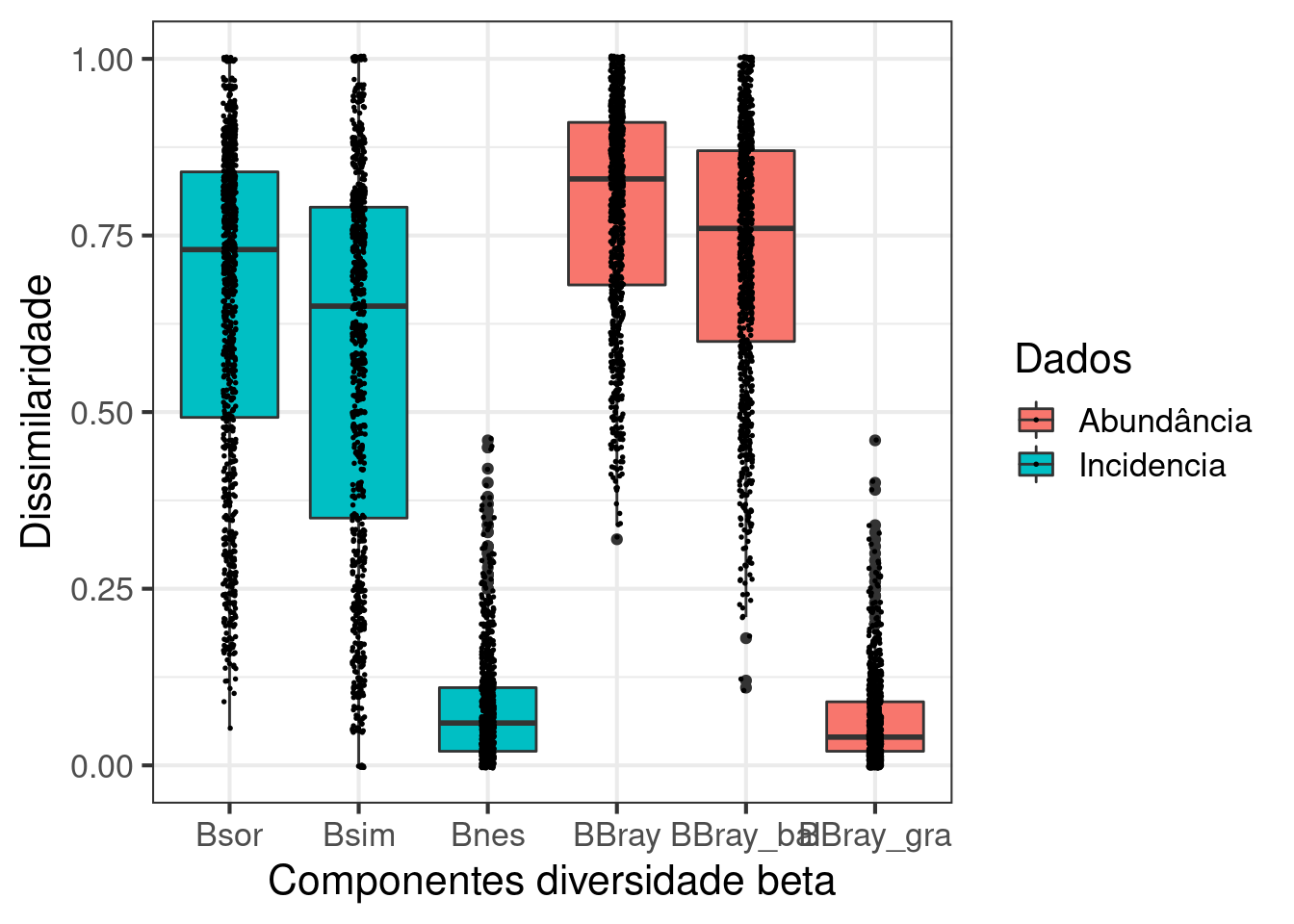
## carregando planilha anuros_ambientais
ambientes <- ecodados::anuros_ambientais
## Distância euclidiana
distancia <- vegdist(ambientes[,c(6,7)], "euclidean")
## criar data frame
dados <- data.frame(round(as.numeric(resultado_PA$beta.sim), 2),
round(as.numeric(distancia), 2))
colnames(dados) <- c("Bsim", "distancia")
## Regressão - Contudo essa não é a melhor análise porque os índices de
# diversidade beta apresenta um platô.
regr <- lm (dados$Bsim ~ dados$distancia)
summary(regr)
#>
#> Call:
#> lm(formula = dados$Bsim ~ dados$distancia)
#>
#> Residuals:
#> Min 1Q Median 3Q Max
#> -0.58919 -0.21205 0.03911 0.19848 0.50133
#>
#> Coefficients:
#> Estimate Std. Error t value Pr(>|t|)
#> (Intercept) 0.4729768 0.0129279 36.59 <2e-16 ***
#> dados$distancia 0.0075119 0.0007092 10.59 <2e-16 ***
#> ---
#> Signif. codes:
#> 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#>
#> Residual standard error: 0.2472 on 944 degrees of freedom
#> Multiple R-squared: 0.1062, Adjusted R-squared: 0.1053
#> F-statistic: 112.2 on 1 and 944 DF, p-value: < 2.2e-16
## Gráfico
ggplot(data = dados, aes(x = distancia*10, y = Bsim)) +
labs(x = "Distância geográfica (km)", y = "Componente substituição diversidade beta") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)