Cap. 13 - Diversidade Filogenética
# Pacotes necessários
library(devtools)
library(ecodados)
library(V.PhyloMaker)
library(vegan)
library(ggplot2)
library(GGally)
library(ggpubr)
library(picante)
library(phytools)
library(ape)
library(geiger)
library(phyloregion)
library(pez)
library(reshape2)
library(betapart)
library(bbmle)
library(vegan)13.1
Carregue os dados - anuros_composicao (i.e., 211 espécies de anuros coletados em 44 localidades na Mata Atlântica), anuros_ambientais (i.e., variáveis climáticas, topográficas e coordenadas geográficas) & filogenia_anuros (filogenia das 211 espécies) - que estão no pacote ecodados. Use a função varpartdo pacote vegan para testar a importância relativa dos efeitos da precipitação anual, range altitudinal e temperatura anual na distribuição espacial da diversidade filogenética (PD) e Endemismo filogenético (PE). Calcule o SES para verificar se os resultados da diversidade filogenética (PD) diferem do esperado ao acaso devido ao número de espécies em cada comunidade. Qual a sua interpretação sobre os resultados?
Solução:
# Carregar as planilhas com os dados
anuros <- ecodados::anuros_composicao
anuros_t <- t(anuros) # Transpor dados
ambiente <- ecodados::anuros_ambientais
filogenia <- ecodados::filogenia_anuros
## Conferir os nomes das espécies
name.check(filogenia, anuros)
#> [1] "OK"
## Colocar os nomes das espécies do data frame na mesma ordem que aparecem na filogenia
anuros_P <- match.phylo.comm(phy = filogenia, comm = anuros_t)$comm
## Phylogenetic diversity (PD)
#*****************************************************************************
anuros_PD <- pd(anuros_P, filogenia)
## Criar data frame
dados <- data.frame(anuros_PD$PD, anuros_PD$SR, ambiente$Prec_anual,
ambiente$Range_altitude, ambiente$Temp_anual)
colnames(dados) <- c("PD", "SR", "Prec_anual", "Range_altitude", "Temp_anual")
## Partição da variância
particao_PD <- varpart(dados$PD, dados$Prec_anual,
dados$Range_altitude, dados$Temp_anual)
plot (particao_PD, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Temperatura'),
bg = c('navy', 'darkorange', 'green'))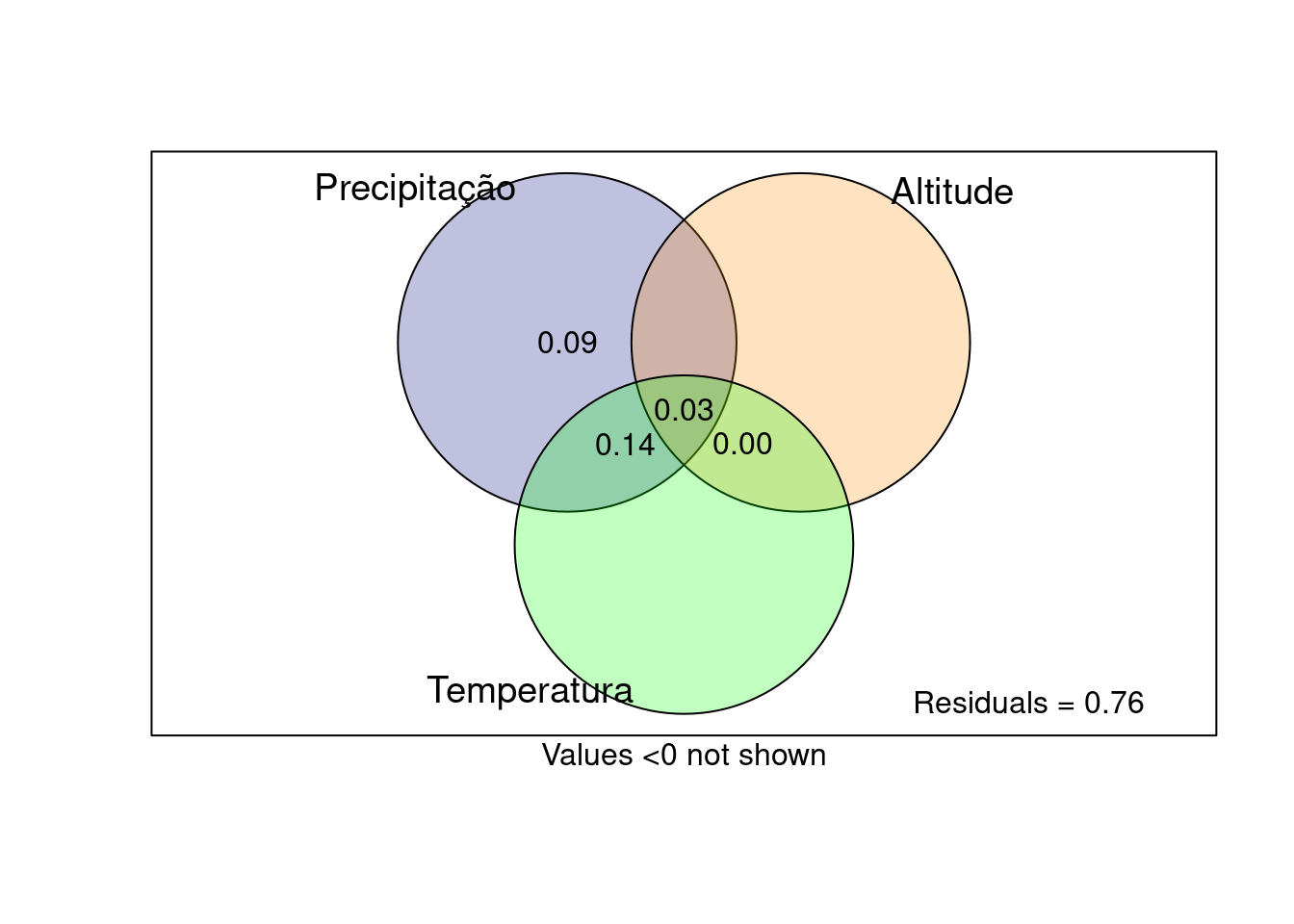
## Gráfico
ggplot(data = dados, aes(x = Prec_anual, y = PD)) +
labs(x = "Precipitação anual (mm)", y = "Diversidade filogenética (PD)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)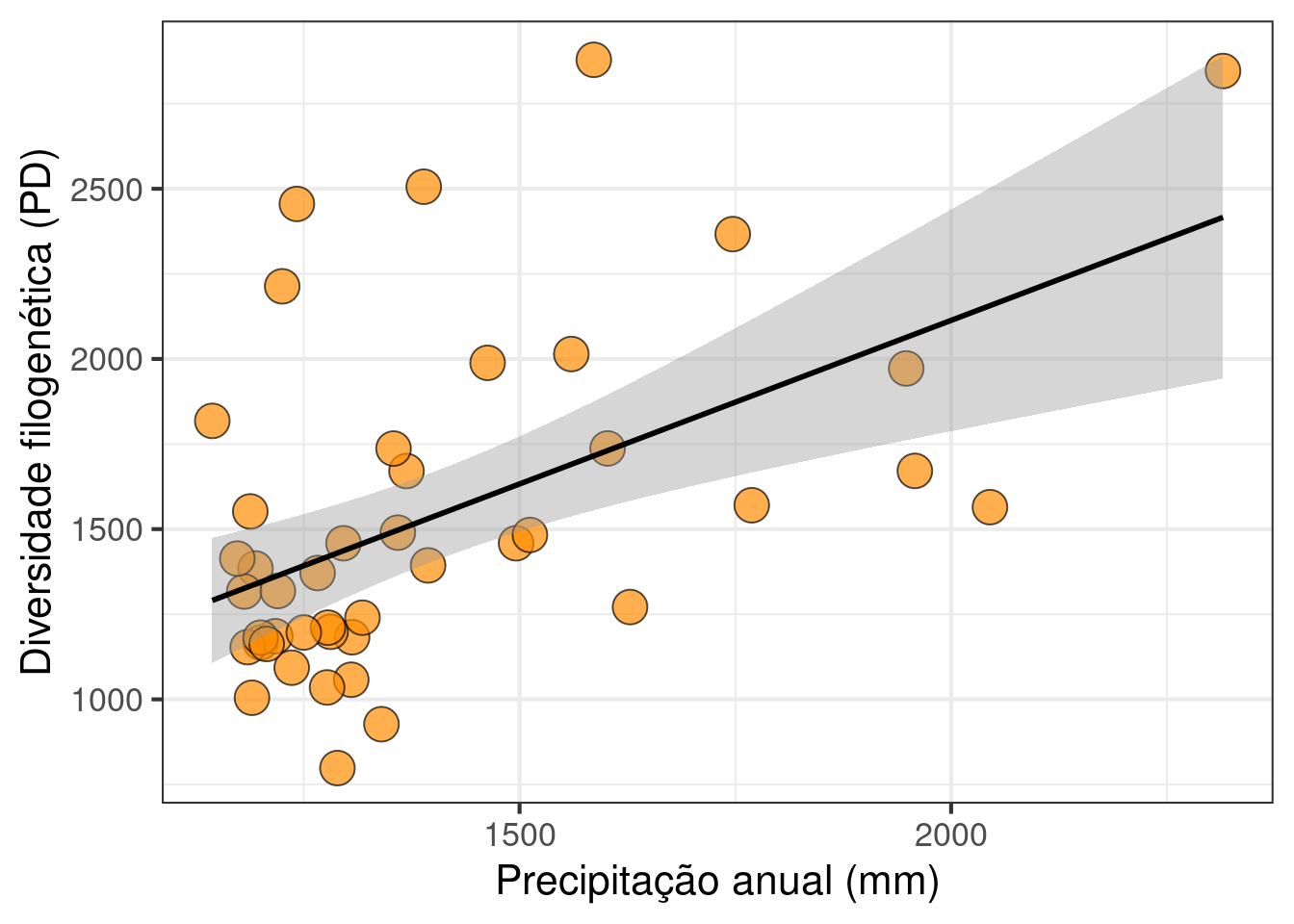
## Phylogenetic Endemism (PE)
#***************************************************************************
# Transformando data.frame em matriz.
anuros_matriz <- as.matrix(anuros_P)
anuros_PE <- phylo_endemism(anuros_matriz, filogenia, weighted = TRUE)
## Adicionando no data frame
dados$PE <- anuros_PE
## Partição da variância
particao_PE <- varpart(dados$PE, dados$Prec_anual,
dados$Range_altitude, dados$Temp_anual)
plot (particao_PE, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Temperatura'),
bg = c('navy', 'darkorange', 'green'))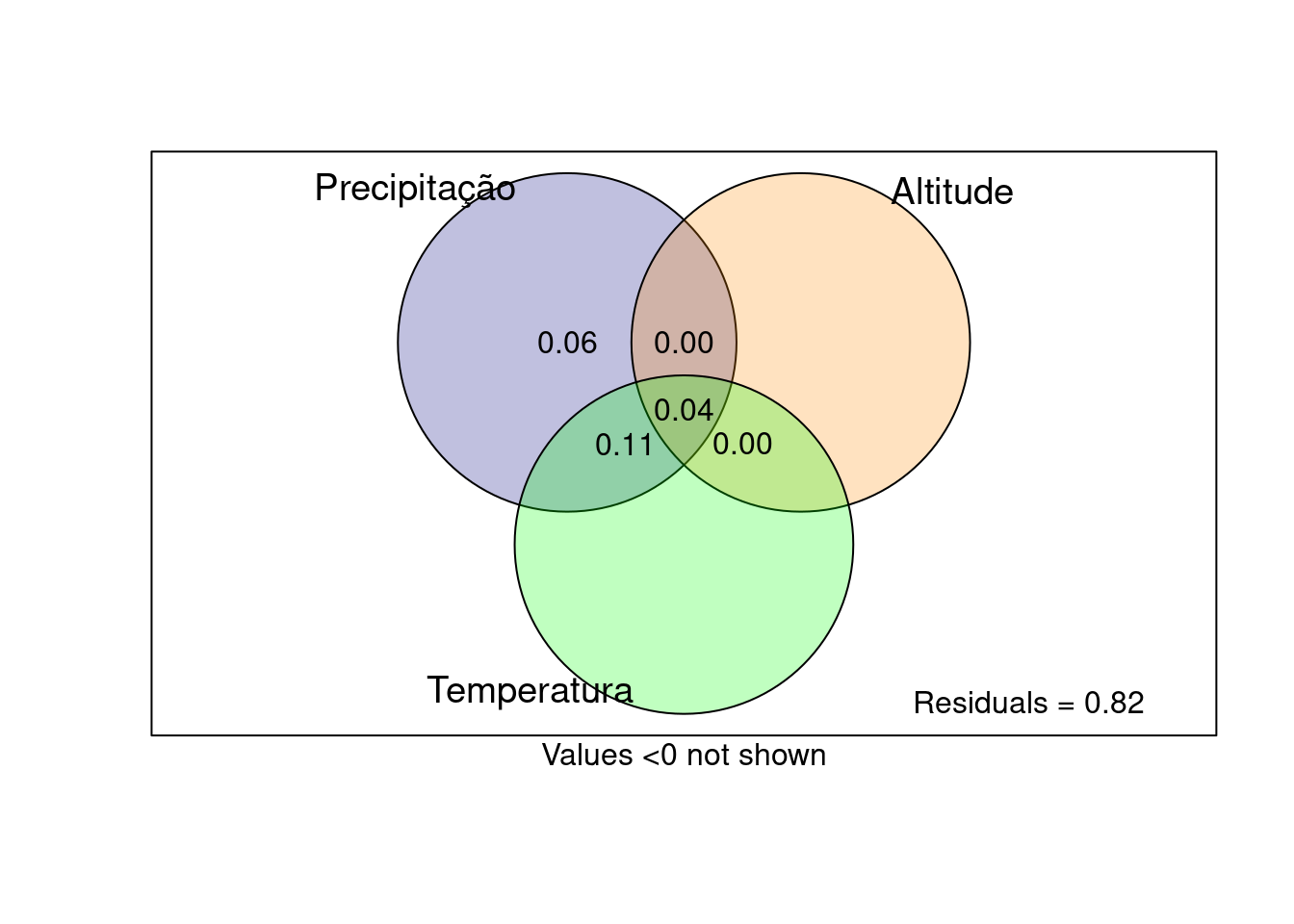
## Gráfico
ggplot(data = dados, aes(x = Prec_anual, y = PE)) +
labs(x = "Precipitação anual (mm)", y = "Endemismo filogenético (PE)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)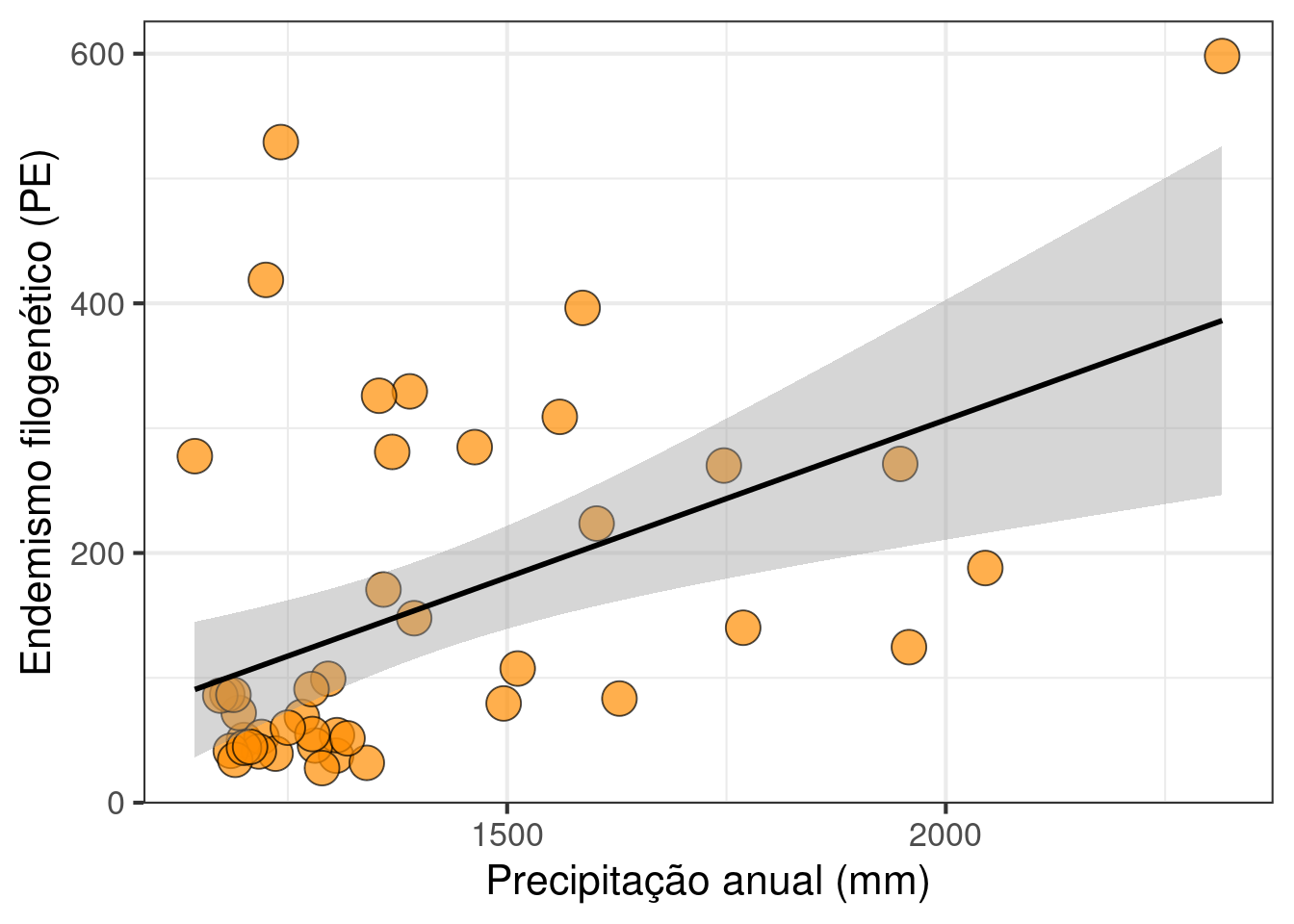
## Standardized Effect Size (SES) para o PD
#*****************************************************************************
resultados_SES_PD <- ses.pd(anuros_P, filogenia,
null.model = "independentswap",
runs = 999)
## Adicionando no data frame
dados$SES <- resultados_SES_PD$pd.obs.z
## Partição da variância
particao_SES <- varpart(dados$SES, dados$Prec_anual,
dados$Range_altitude, dados$Temp_anual)
plot (particao_SES, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Temperatura'),
bg = c('navy', 'darkorange', 'green'))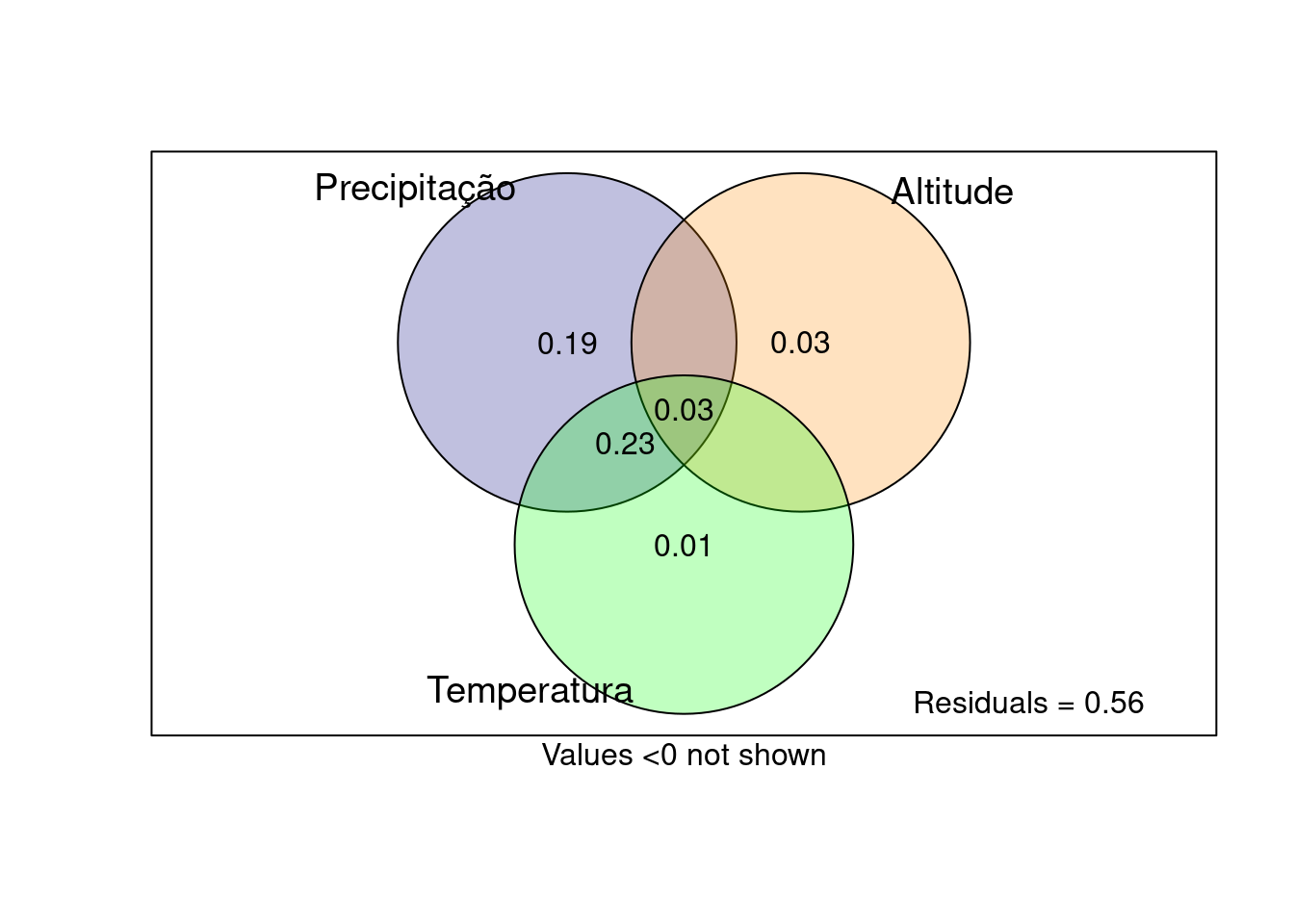
## Gráfico
ggplot(data = dados, aes(x = Prec_anual, y = SES)) +
labs(x = "Precipitação anual (mm)", y = "Standardized Effect Size (PD)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)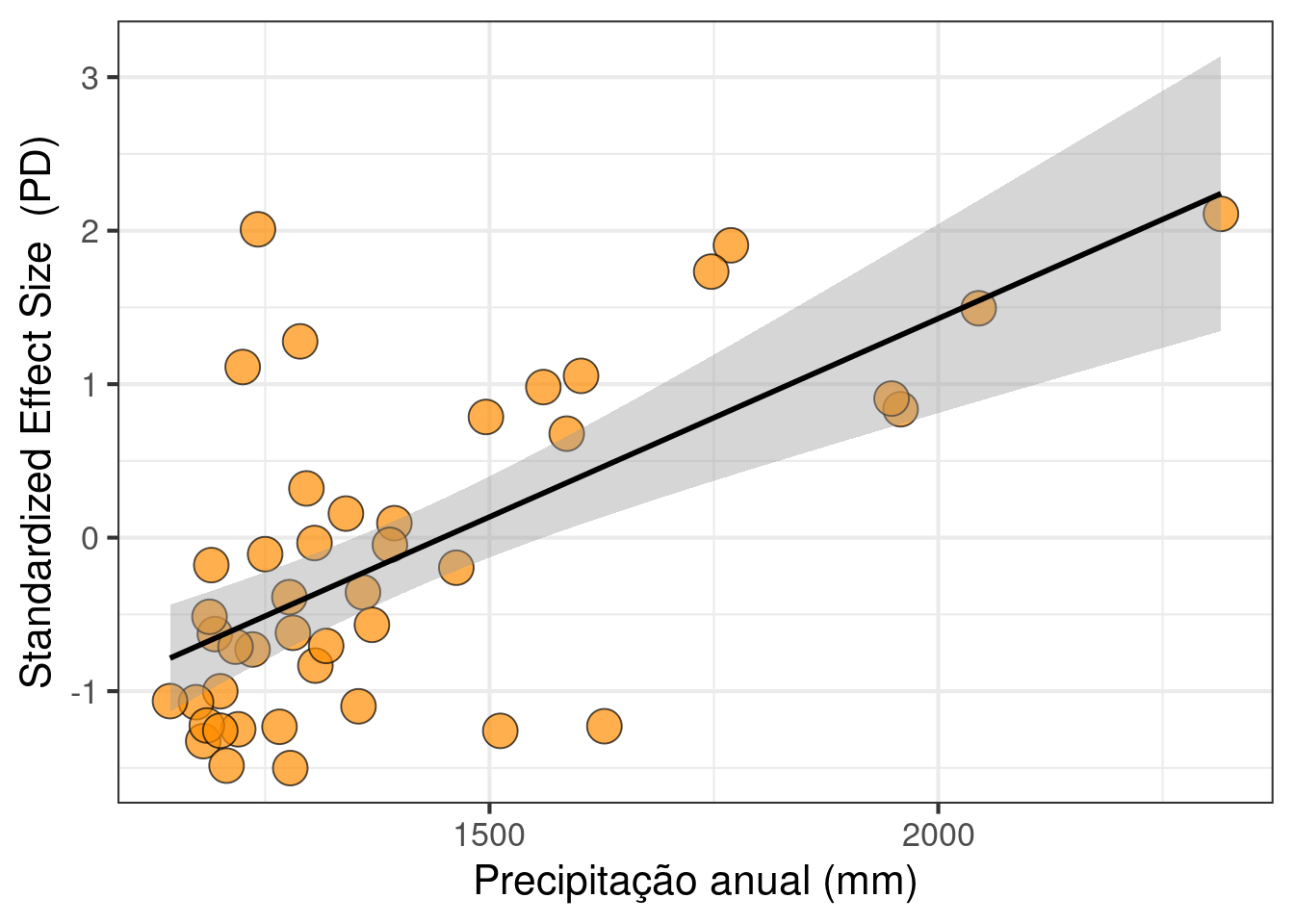
13.2
Carregue os dados - anuros_composicao (i.e., 211 espécies de anuros coletados em 44 localidades na Mata Atlântica), anuros_ambientais (i.e., variáveis climáticas, topográficas e coordenadas geográficas) & filogenia_anuros (filogenia das 211 espécies) - que estão no pacote ecodados. Use a função varpartdo pacote vegan para testar a importância relativa dos efeitos da precipitação anual, range altitudinal e temperatura anual na distribuição espacial do NRI e NTI. Qual a sua interpretação sobre os resultados?
Solução:
## Nearest Relative Index (NRI)
#*****************************************************************************
resultados_NRI <- ses.mpd(anuros_P, cophenetic(filogenia),
null.model = "taxa.labels",
abundance.weighted = FALSE,
runs = 999)
## Inserir no data frame
dados$NRI <- -1*(resultados_NRI$mpd.obs.z)
## Partição da variância
particao_NRI <- varpart(dados$NRI, dados$Prec_anual,
dados$Range_altitude, dados$Temp_anual)
plot (particao_NRI, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Temperatura'),
bg = c('navy', 'darkorange', 'green'))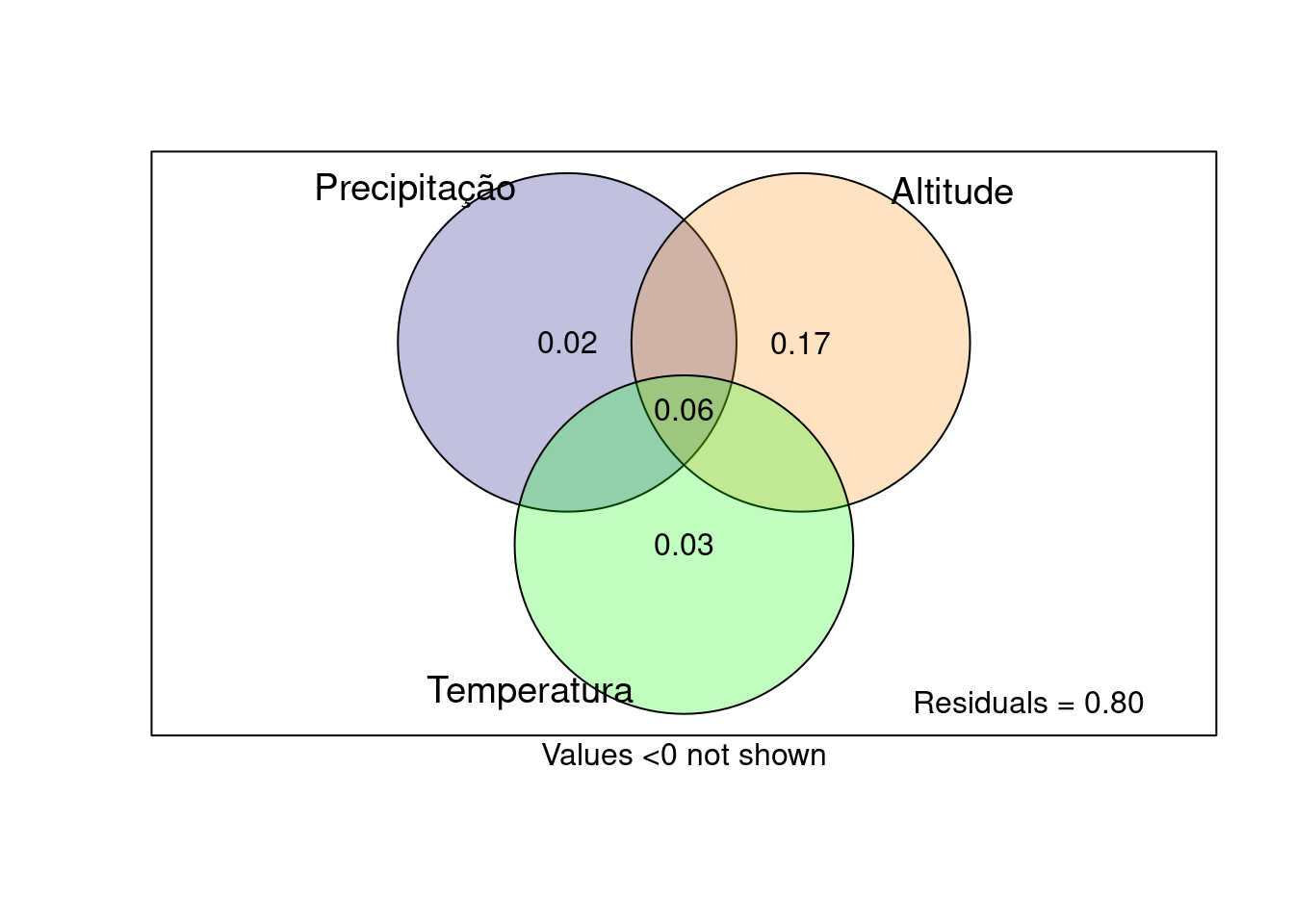
## Gráfico
ggplot(data = dados, aes(x = Range_altitude, y = NRI)) +
labs(x = "Range altitudinal (m)", y = "Nearest Relative Index (NRI)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)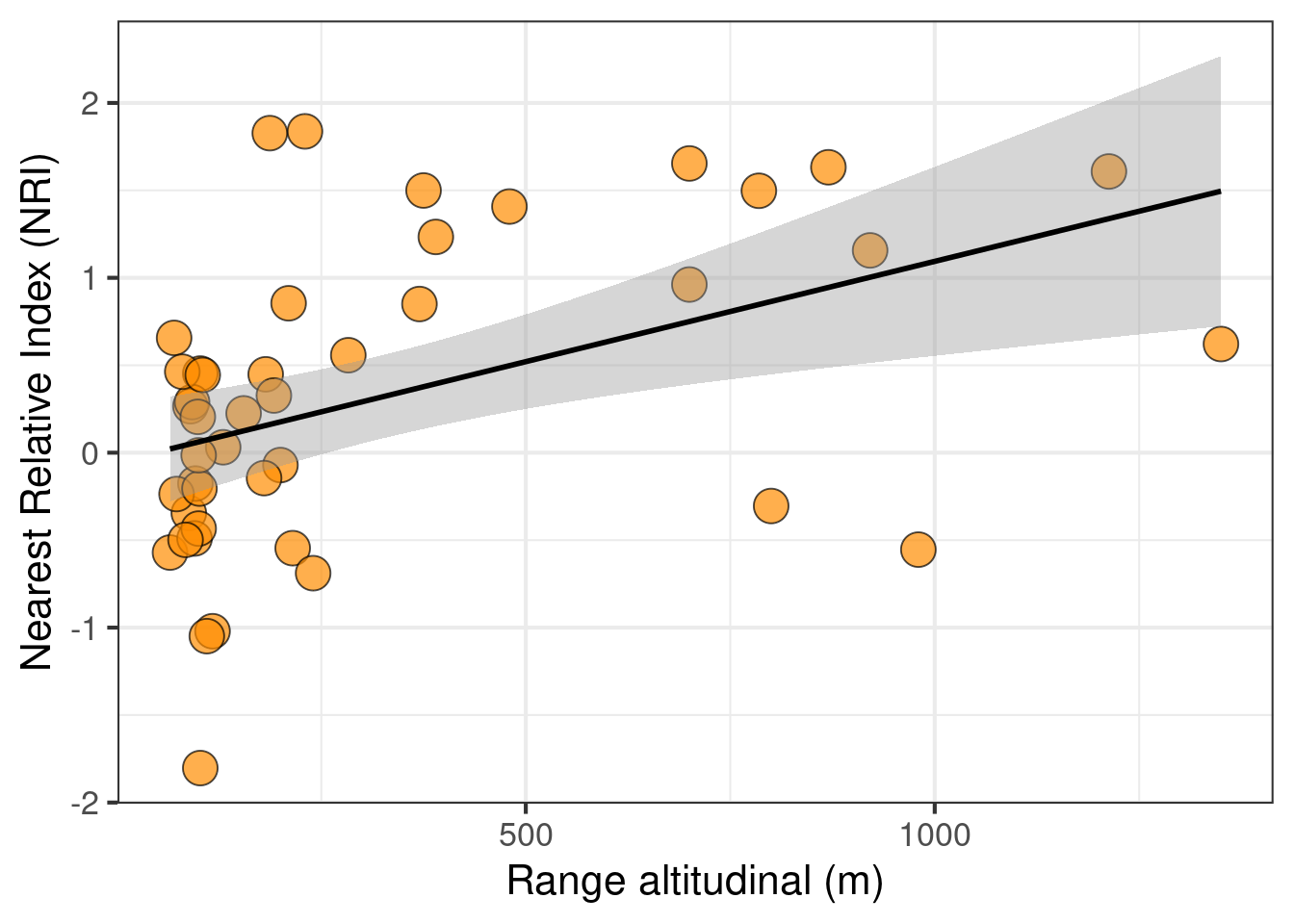
## Nearest Taxon Index (NTI)
#*****************************************************************************
resultados_NTI <- ses.mntd(anuros_P, cophenetic(filogenia),
null.model = "taxa.labels",
abundance.weighted = FALSE,
runs = 999)
## Inserir no data frame
dados$NTI <- -1*(resultados_NTI$mntd.obs.z)
## Partição da variância
particao_NTI <- varpart(dados$NTI, dados$Prec_anual,
dados$Range_altitude, dados$Temp_anual)
plot (particao_NTI, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Temperatura'),
bg = c('navy', 'darkorange', 'green'))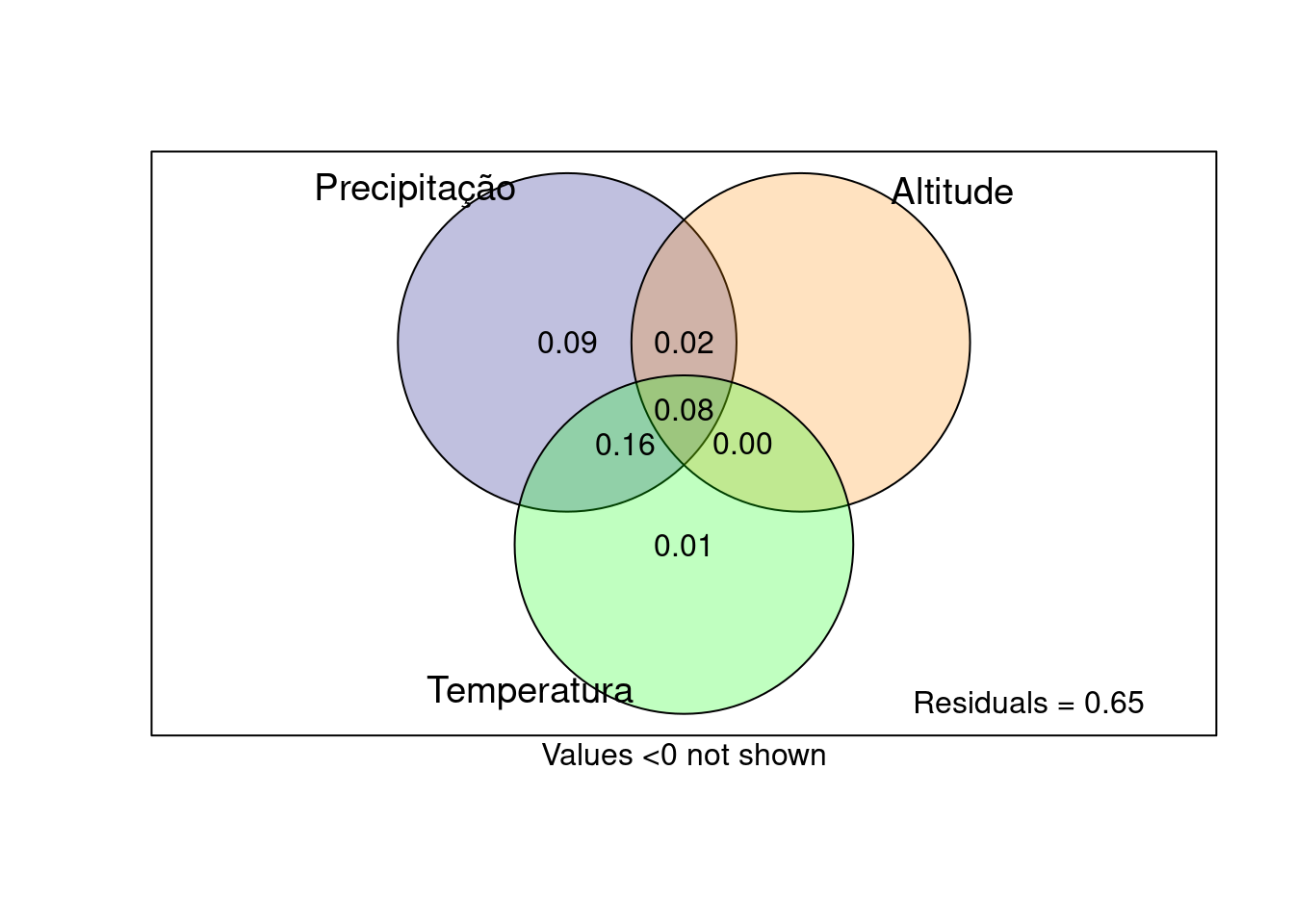
## Gráfico
ggplot(data = dados, aes(x = Range_altitude, y = NTI)) +
labs(x = "Precipitação anual (mm)", y = "Nearest Taxon Index (NTI)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)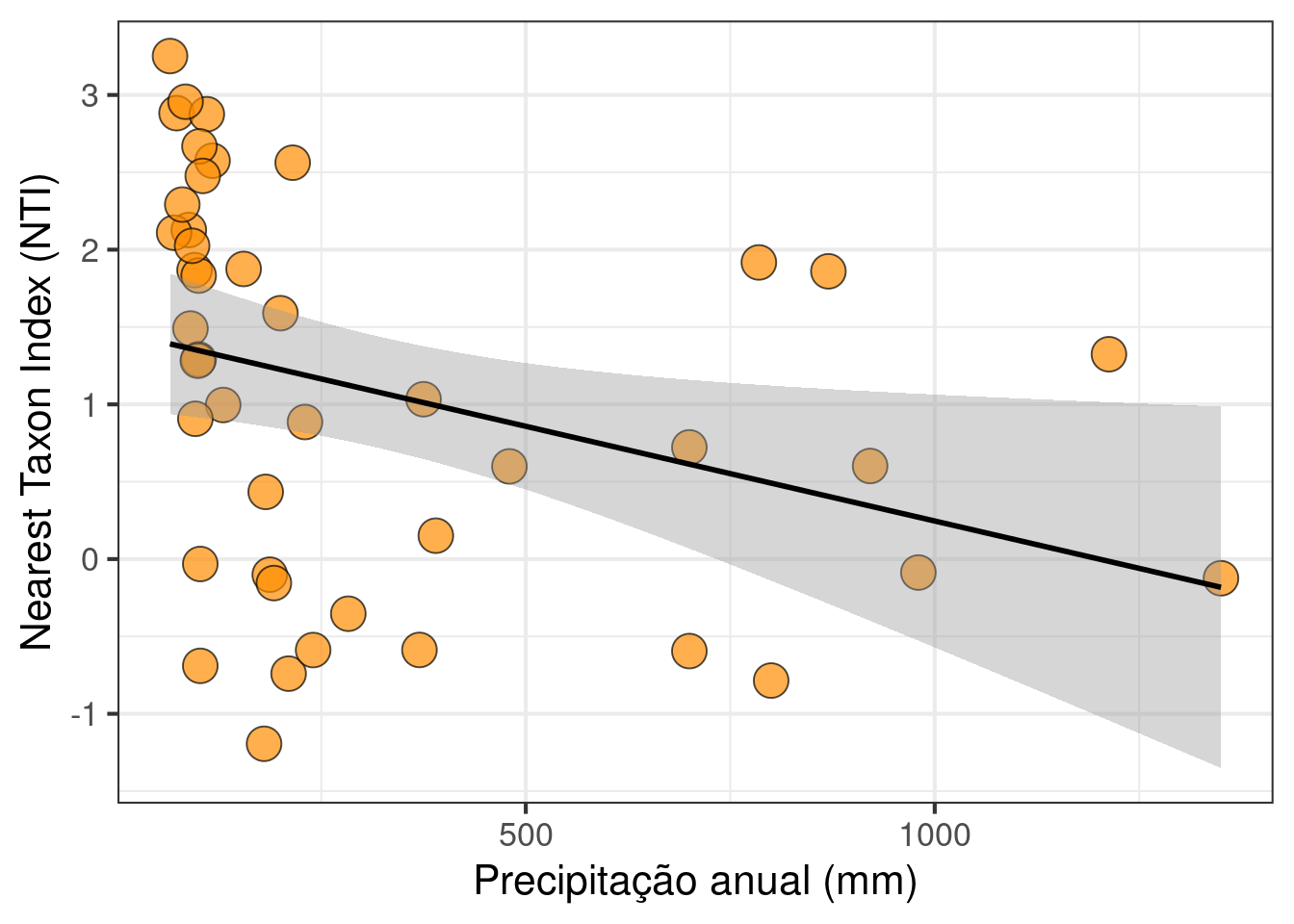
13.3
Carregue os dados - anuros_composicao (i.e., 211 espécies de anuros coletados em 44 localidades na Mata Atlântica), anuros_ambientais (i.e., variáveis climáticas, topográficas e coordenadas geográficas) & filogenia_anuros (filogenia das 211 espécies) - que estão no pacote ecodados. Use a função varpartdo pacote vegan para testar a importância relativa dos efeitos da precipitação anual, range altitudinal e distância geográfica na distribuição espacial dos diferentes componentes da diversidade beta filogenética (Phylosor). Qual a sua interpretação sobre os resultados?
Solução:
# Temos que transformar os dados para presença e ausência das espécies nas comunidades.
anuros_PA <- decostand(anuros_P, "pa")
## Phylosor
#*****************************************************************************
# Partição dos componentes do Phylosor.
resultados_Phylosor <- phylo.beta.pair(anuros_PA,
filogenia,
index.family = "sorensen")
## vamos calcular a similaridade entre as variáveis ambientais
prec_dist <- vegdist(ambiente$Prec_anual, "euclidian")
alt_dist <- vegdist(ambiente$Range_altitude, "euclidian")
coord_dist <- vegdist(ambiente[,c(5,6)], "euclidian")
# Vamos preparar os dados .
dados_phylosor <- data.frame(
substituicao = as.numeric(resultados_Phylosor$phylo.beta.sim),
aninhamento = as.numeric(resultados_Phylosor$phylo.beta.sne),
sorensen = as.numeric(resultados_Phylosor$phylo.beta.sor),
dis_prec = as.numeric(prec_dist),
dis_alt = as.numeric(alt_dist),
dis_coord = as.numeric(coord_dist))
## Partição da variância do componente substituição do Phylosor
#*******************************************************************************
particao_subst <- varpart(dados_phylosor$substituicao, dados_phylosor$dis_prec,
dados_phylosor$dis_alt, dados_phylosor$dis_coord)
plot (particao_subst, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Distância'),
bg = c('navy', 'darkorange', 'green'))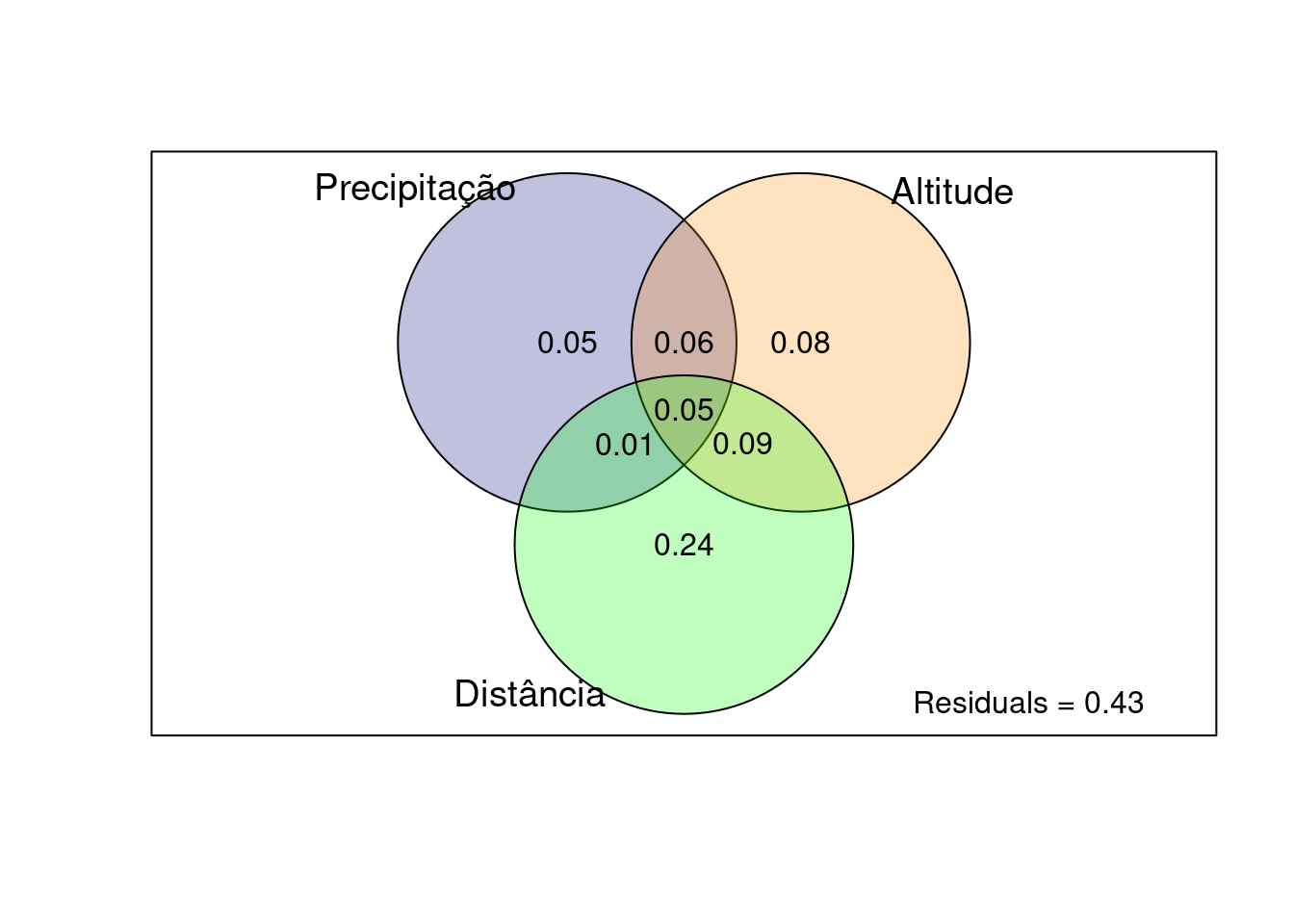
## Gráfico
ggplot(data = dados_phylosor, aes(x = dis_coord*10, y = substituicao)) +
labs(x = "Distância geográfical (km)",
y = "Componente substituição da diversidade beta \n filogenética (Phylosor)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)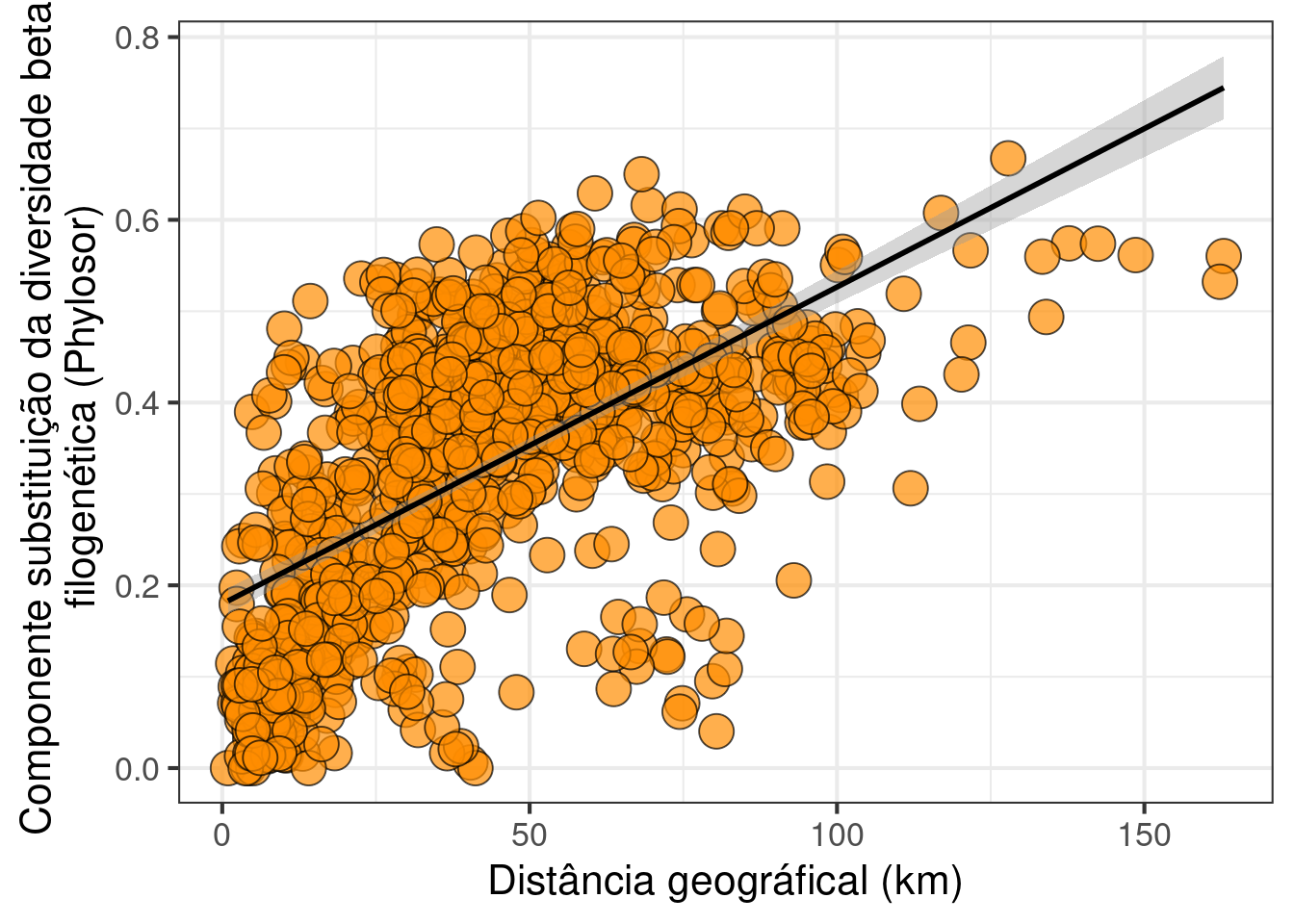
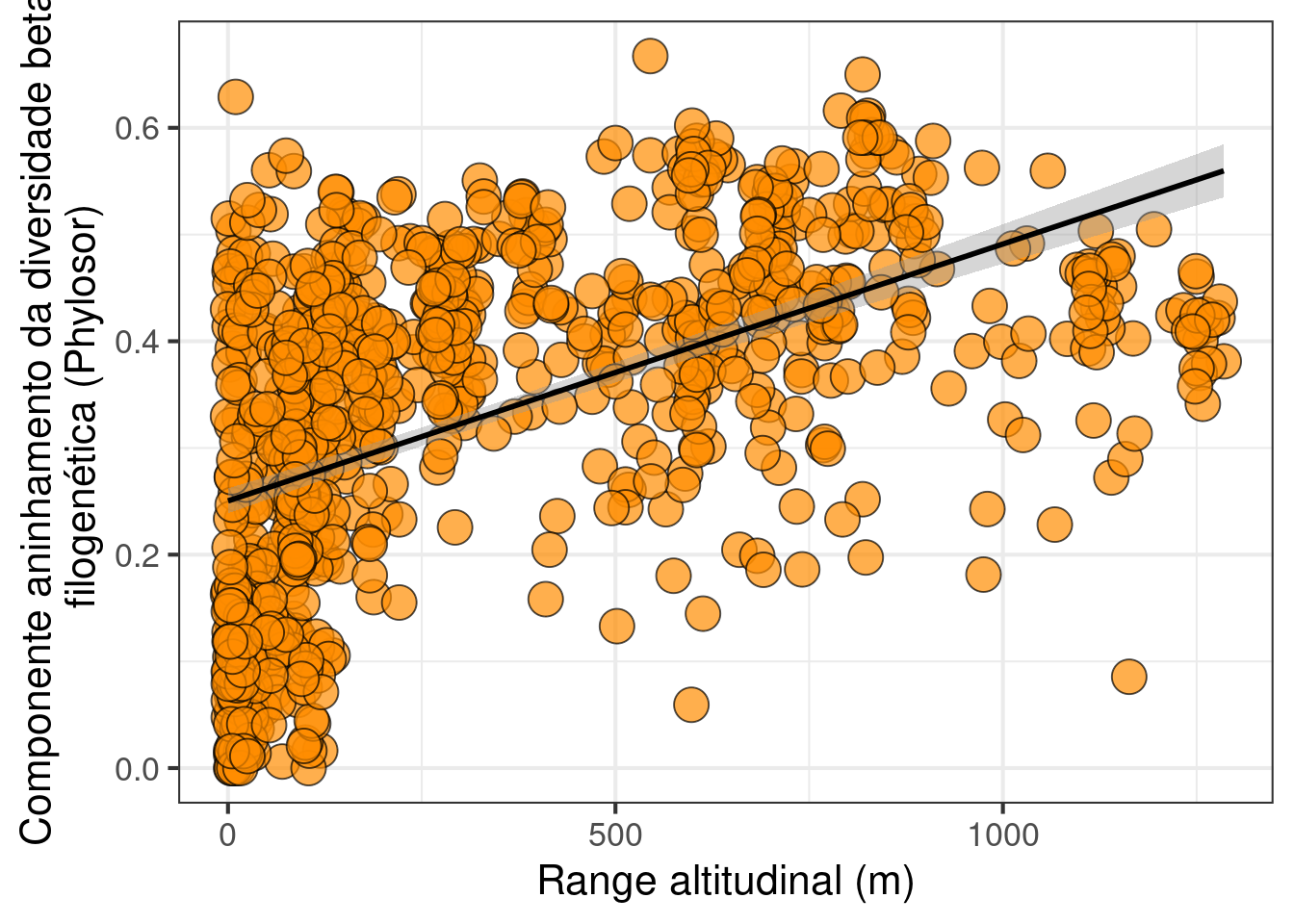
## Partição da variância do componente aninhamento do Physolor
#*******************************************************************************
particao_anin <- varpart(dados_phylosor$aninhamento, dados_phylosor$dis_prec,
dados_phylosor$dis_alt, dados_phylosor$dis_coord)
plot (particao_anin, digits = 1, Xnames = c('Precipitação', 'Altitude', 'Distância'),
bg = c('navy', 'darkorange', 'green'))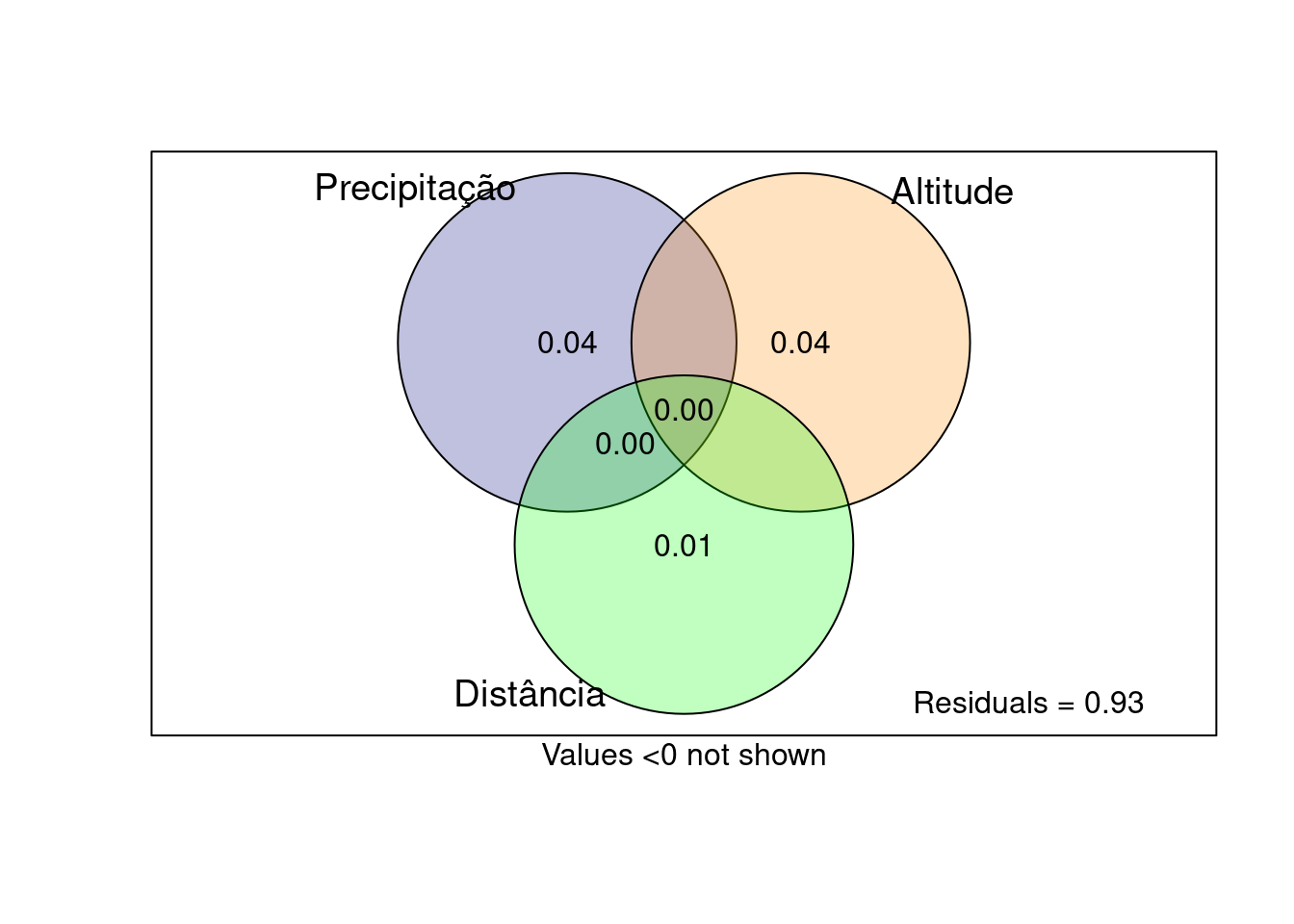
## Gráfico
ggplot(data = dados_phylosor, aes(x = dis_alt, y = substituicao)) +
labs(x = "Range altitudinal (m)",
y = "Componente aninhamento da diversidade beta \n filogenética (Phylosor)") +
geom_point(size = 6, shape = 21, fill = "darkorange", alpha = 0.7) +
theme(legend.position = "none") +
geom_smooth(method = lm, se = TRUE, color = "black") +
theme_bw(base_size = 16)